La etiología del TDAH es compleja y multifactorial. La teoría más plausible hasta la fecha es que surge de múltiples factores de riesgo genéticos y ambientales, que tienen pequeños efectos individuales y actúan en conjunto para aumentar la suscep- tibilidad a desarrollar el trastorno. Esto constituye un patrón de herencia no mendeliana, compleja, con posible penetrancia incompleta y expresividad variable, que sugiere la acción conjunta de múltiples genes de efecto moderado o discreto con fac- tores ambientales.
En este artículo se realiza una revisión narrativa del tema, teniendo en cuenta la descripción de genes candidatos y estudio de transcriptomas y GWAS.
Palabras clave
TDAH – Genética – GWAS – Transcriptoma – Herencia.
Angemi JA. “Genética del trastorno por déficit de atención e hiperactividad”. Psicofarmacología 2024;135:18-30. Puede consultar otros artículos publicados por los autores en la revista Psicofarmacología en sciens.com.ar
La etiología del TDAH (Trastorno por déficit de atención e hiperactividad) es compleja y multifactorial. La teoría más plausible hasta la fecha es que surge de múltiples factores de riesgo genéticos y ambientales, que t ienen pequeños efectos individuales y actúan en conjunto para aumentar la suscepti- bilidad a desarrollar el trastorno (1) (Ver Cuadro 1).
Metaanálisis de múltiples estudios de gemelos a gran esca- la estiman una heredabilidad del 70% al 80% para el TDAH infantil (2) y alrededor del 70% para el TDAH clínicamente diagnosticado en adultos (3).
Además, las tasas de concordancia en gemelos monocigóti- cos son consistentemente más altas que las de los gemelos di- cigóticos, alrededor de 80% y 40%, respectivamente (4). Los estudios de adopción sugieren que los factores familiares del TDAH son atribuibles a factores genéticos más que a factores ambientales compartidos. Las tasas de TDAH entre parientes biológicos de niños con TDAH adoptados son mayores que entre parientes adoptivos de esos niños (5).
La heredabilidad no varía entre varones y mujeres y entre los distintos subtipos (inatento e hiperactivo/ impulsivo) (6).
Estudios en gemelos demuestran que en la genética del TDAH existen factores estables y dinámicos que influyen a lo largo del desarrollo desde la niñez hasta la edad adulta temprana. El componente estable del riesgo genético sugiere que el TDAH persistente y su forma pediátrica están genéti- camente relacionados. El componente dinámico sugiere que el conjunto de variantes genéticas que explican la aparición del TDAH difiere de aquellas que explican la persistencia y remisión del trastorno (3).
En cuanto a la comorbilidad, que es la regla y no la excep- ción, un factor genético compartido latente representa hasta el 45% de la covarianza en los síntomas de externalización, internalización y fobia de la infancia y el 31% de la covarianza en los síntomas del neurodesarrollo infantil (7). La correlación entre TDAH y coeficiente intelectual (CI) se explica en un 91% por factores genéticos (3).
La contribución de estudios genéticos realizados sobre todo a partir de la década del 1990, resultan fundamentales para entender la complejidad de las interacciones gen-ambiente (GxA), manifestación clínica y abordaje terapéutico del TDAH. Se remite al Cuadro 2 para ver definiciones basadas en el Na- tional Human Genome Research de USA (www.genome.gov), que facilitarán la lectura del presente artículo.
La variación epigenética se clasifica en modificaciones he- reditarias y dependientes del contexto, que ocurren en células germinales y se transmiten de generación en generación, o en células somáticas y persisten dinámicamente solo a lo largo de la vida (no se transmite a la descendencia).
El nucleosoma es la subunidad fundamental de la estruc- tura de la cromatina y comprende un tramo corto de ADN envuelto alrededor de dos copias de las histonas centrales H2A, H2B, H3 y H4.
Las modificaciones postraduccionales de las proteínas his- tonas, predominantemente la acetilación y la fosforilación, así como la metilación del ADN, regulan la arquitectura de la cromatina, lo que afecta la unión del factor de transcripción a secuencias específicas de ADN y, en última instancia, la expresión genética. La acetilación y fosforilación de histonas pueden facilitar la deposición o eliminación de modificacio- nes más estables de la cola de histonas, como la metilación, y por lo tanto tienen el potencial de moderar la expresión ge- nética a largo plazo. Finalmente, estos mecanismos se com- plementan con la represión postranscripcional acelerada por miARN que se unen a las 3’-UTR de los ARNm objetivo para regular su expresión ya sea reprimiendo la traducción o indu- ciendo la degradación del ARNm (8).
Cuadro 1
Factores de riesgo ambientales en el TDAH
| Prematuridad |
| Exposición intraútero a tabaco, alcohol, marihuana, paracetamol, cafeína, Pb, Hg. (¿pesticidas y agroquímicos?) |
| Complicaciones pre y perinatales |
| Traumatismo cerebral |
| Deprivación socioeconómica |
| Disrupción familiar |
| Falta de criterios educativos |
Los factores estresantes prenatales afectan el riesgo de ano- malías cognitivas y conductuales asociadas con el TDAH. La evidencia indica la participación de alteraciones en la mieliniza- ción y la transmisión GABAérgica. En ratones, las crías nacidas de madres expuestas a estrés por restricción prenatal durante el embarazo mostraron hiperactividad locomotora y déficits en la atención, el procesamiento de la información, el aprendizaje y la memoria, así como en la interacción social (8, 9).
Los fenotipos cerebrales alterados en el TDAH se han con- siderado endofenotipos clave para el trastorno, y la investiga- ción de las influencias genéticas en estas medidas constituye un aporte importante para entender la correlación.
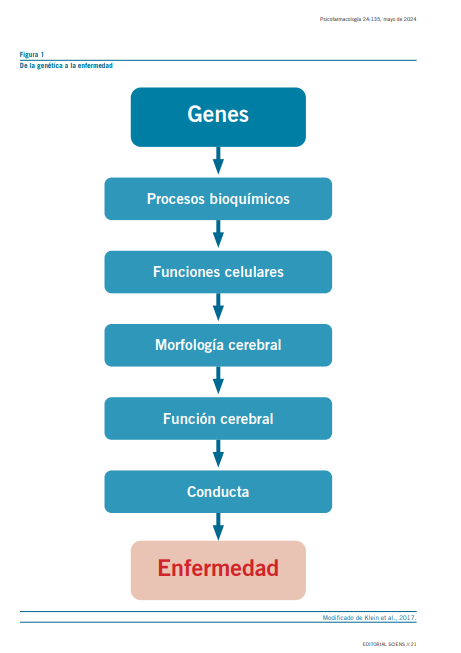
En sus inicios, los estudios genéticos se enfocaron en genes candidatos en animales de acuerdo al mecanismo de acción de fármacos en el tratamiento del trastorno (sistemas dopa- minérgico, catecolaminérgico y serotoninérgico). Posterior- mente se enfocó sobre funciones afectadas (atención, control de impulsos, funciones ejecutivas) y desarrollo del sistema nervioso, la morfogénesis de la proyección neuronal, la comu- nicación entre células, la señalización de receptores/sinapsis glutamatérgicas, el desarrollo de organismos multicelulares o las proyecciones neuronales y los componentes sinápticos. Son de mucha utilidad los correlatos con neuroimágenes es- tructurales y funcionales, y con estudios neuropsicológicos
(10) (Ver Figura 1) (11).
Zhou et al., (2008) aplicaron el metaanálisis de GWAS, en- contrando un vínculo significativo en todo el genoma para una región del cromosoma 16 entre 64 Mb y 83 Mb. Esta resulta la evidencia más fuerte de localización cromosómica (12).
Estudios de GWAS establecen una heredabilidad de SNP en un rango de 0,1 a 0,28, apoyando la contribución de variantes comunes a la etiología del TDAH. Un importante GWAS, que comprendió 20.000 pacientes con TDAH y 35.000 controles, identificó 12 locus de riesgos independientes, agregando nue- va información importante sobre la biología subyacente del TDAH. La heredabilidad se calcula como el cociente de la co- rrelación calculada y el coeficiente de relación. Esto significa que la correlación esperada máxima entre padres e hijos es de 0,5. Si el coeficiente de correlación es mayor, significa que la similitud es causada por otros factores no genéticos (13).
Genes candidatos
Se trata de los genes que codifican los transportadores de dopamina (DA) y serotonina (5HT), SLC6A3/DAT1 y SL- C6A4/5HTT, los genes que codifican los receptores de do- pamina D4 y D5, (DRD4 y DRD5), el receptor de serotonina
HTR1B, y el gen de la proteína 25 asociada a sinaptosomas, SNAP25. Algunos genes adicionales (que codifican la DA beta-hidroxilasa [DBH], el adrenoceptor alfa 2A [ADRA2A], la triptófano hidroxilasa 2 [TPH2] y la monoaminooxidasa A [MAOA) se encontraron sugestivamente asociados con el TDAH en los metaanálisis.
Una muestra europea de 674 familias con probandos de tipo combinado de TDAH, recopilada para el proyecto Interna- tional Multisite ADHD Gene (IMAGE), identificó asociaciones con genes candidatos para el TDAH, como ADRAB2, DAT1, DRD4, TPH2 y MAOA (11, 14).
A continuación se describen los más significativos.
Gen SLC6A3, DAT1
Localizado en cromosoma 5p.15.3.2, codifica para una proteína transportadora de solutos (DAT), responsable de la recaptación de dopamina desde la hendidura sináptica hacia la neurona presináptica, lo que representa un mecanismo pri- mario de regulación de la disponibilidad de DA en el espacio sináptico. El polimorfismo más estudiado es un número varia- ble de secuencias repetidas en tándem (VNTR) en la región 3’ no traducida (3’UTR) que tiene 40 pares de bases (pb) de longitud. Los alelos más comunes son aquellos con 9 y 10 repeticiones (9R y 10R), el primero relacionado al TDAH de la infancia y el segundo al TDAH persistente (11, 15).
Un grupo brasilero realizó una investigación de un VNTR de seis copias de 30 pb ubicado en el intrón 83 de SLC6A3 en una muestra compuesta por 94 pacientes adultos con TDAH (utilizando criterios del DSM-IV) y 481 sujetos de control. En- tre los 94 pacientes con TDAH, 58 (61,20%) eran hombres y 36 (38,30%) eran mujeres, mientras que, en el grupo de con- trol, 323 (67,15%) eran hombres y 158 (32,85%) eran mu- jeres. Las edades medias de los grupos con TDAH y de control fueron, respectivamente, 33,0 (sd = 9,21) y 32,5 (sd = 9,5). Encontraron una fuerte asociación con TDAH (0.05) (16).
Pacientes homocigotos para el alelo SLC6A3/DAT1 10R y portadores del alelo DRD4 7R exhibieron una menor ocupa- ción de DAT después del tratamiento con MPH en el núcleo caudado y el putamen derecho e izquierdo (17).
Los modelos animales en ratones KO para el gen son de los más conocidos. La conducta alterada en estos animales mejora con anfetamina, MPH y atomoxetina (18).
Los ratones DAT-KO presentan alteración de la función del autorreceptor DA y muestran una disminución significativa de la expresión de la proteína tirosina hidroxilasa (TH), a pesar
Cuadro 2
Definiciones útiles sobre genética
| SNPs (Polimorfismos de nucleó- tido único) | Variación de una secuencia de DNA que ocurre cuando se altera un solo nucleótido (adenina, timina, citosina o guanina) de la secuencia de referencia. Los SNP suelen presentarse en al menos el 1 % de la población y son el tipo más común de variación genética en las personas. Sirve para detectar riesgo de ciertas enfermedades, de biomarcadores para ayudar a predecir el riesgo de algunas afecciones o para predecir la respuesta de una persona a determinados medicamentos. También se usan para rastrear la herencia de variantes que causan enferme- dades en estudios familiares. |
| GWAS (Estudio de asociación de genoma completo) | Estudio en el que se comparan los marcadores de ADN en el genoma completo de las perso- nas con una enfermedad o rasgo con el de las personas que no tienen la enfermedad o rasgo. Una vez que se han identificado esas variantes genómicas, por lo general se usan para buscar variantes cercanas que contribuyan directamente a la enfermedad o rasgo. |
| Transcriptoma | Totalidad de todas las lecturas de genes presentes en una célula o tejido, establecidas por el conjunto de moléculas de mARN y de ARN no-codificante presentes. |
| TWAS (Transcriptome-wide association study) | Estudio de asociación de todo el transcriptoma. Se utiliza para la identificación y priorización de genes causales candidatos en el análisis de genes candidatos después de un estudio de GWAS. |
| LD (Linkage disequilibrium) | Desequilibrio de ligamiento. Se produce cuando hay alelos (marcadores de ADN) que debido a su cercanía física en un cromosoma se presentan juntos de manera más frecuente de lo que se esperaría por azar. |
| TDT (Transmission- disequilibrium test) | Prueba de LD basada en estudios de familias que brinda una fuerte probabilidad de estable- cer el vínculo entre alelos y fenotipos que puede ser causal (es decir, el locus marcador es el alelo de la enfermedad/rasgo) o se debe a un desequilibrio de ligamiento. La TDT equivale a un experimento aleatorio. |
| EWAS (Epigenome-wide associa- tion studies) | Examen de un conjunto de marcas epigenéticas cuantificables en todo el genoma, especial- mente la metilación del ADN, en diferentes individuos, para establecer asociaciones entre la variación epigenética y un fenotipo o rasgo. |
| VNTR (Variable Number of Tandem Repeats) | Secuencias de ADN repetitivas y adyacentes (en “tándem”) que varían entre individuos. Son marcadores genéticos útiles para la identificación y el estudio de enfermedades genéticas, así como en la investigación forense y de paternidad. Su longitud puede variar entre 10 y 100 pares de bases. |
| PRS (Puntuación de riesgo poligénico) | Uso de información genómica para evaluar las probabilidades de que una persona tenga o desarrolle una afección médica particular. La PRS de una persona es un cálculo estadístico que se basa en la presencia o ausencia de múltiples variantes genómicas, sin tomar en cuenta factores ambientales u otros factores. |
| Microarrays | Técnica que estudia la expresión de muchos genes a la vez. Consiste en colocar miles de secuencias génicas (ADN o zARN) sobre un chip y cuantificar los apareamientos de bases complementarias. |
de alteraciones no significativas en el número de neuronas TH positivas. (19) El bloqueo de receptores NMDA aumenta los síntomas, e impide el efecto terapéutico de los estimulantes e IRSS. Esto sugiere la participación de las vías glutamatér- gicas cortico-estriatales en los efectos de estos fármacos. Es importante destacar que, mientras que el efecto estimulante psicomotor de los psicoestimulantes se correlaciona con el aumento de los niveles extracelulares de DA en las principales áreas cerebrales de DA en ratones normales, los psicoestimu- lantes inhiben la actividad sin causar una disminución de los niveles elevados de DA en ratones DAT-KO (20).
Gen DRD4
Codifica para el receptor dopaminérgico D4, acoplado a proteína G, ubicado cerca del telómero del cromosoma 11p15.51. Los distintos alelos tienen capacidades diferentes para disminuir los niveles de AMPc, resultando el 4R ser el más eficiente.
Se destaca un estudio realizado a una población chilena de 60 niños de la etnia Rapa Nui con TDAH en la Isla de Pascua, vs 30 controles neurotípicos. En este grupo, se asoció el ge- notipo 2R/4R con el TDAH, de forma similar a lo que sucede en población asiática (de donde proviene su origen). Resulta muy interesante que el mismo equipo halló diferencias en la prevalencia del TDAH en distintos grupos poblacionales, a sa- ber: 8,33% de los escolares de origen aymara, 9,73% de los escolares de Santiago de Chile y 20,79% de los escolares de origen Rapa Nui (21).
El polimorfismo DRD4 más estudiado en el TDAH ha sido el VNTR de 48 pb en el exón 3 del gen, siendo los alelos de 2, 4 y 7 repeticiones los más comunes. Es altamente expresado en corteza orbitofrontal y cingulada anterior (11, 22).
El DRD4 48 pb VNTR parece mostrar una asociación di- ferencial con el TDAH en niños, donde puede ser uno de los factores de riesgo más fuertes entre las variantes genéticas comunes y en adultos, donde no se pudo observar asociación con el trastorno, aunque sí podría ocurrir como parte de inte- racciones gen-ambiente.
Los portadores del alelo 7R mostraron una disminución del volumen de la corteza frontal a una edad más temprana y me- nor respuesta a tratamiento con estimulantes (23).
Los modelos de ratones DRD4-KO resultaron fallidos en de- mostrar síntomas compatibles con TDAH (24).
Gen COMT
Codifica para la enzima Catecol-O-metil-transferasa (ubica- do en 22q11.21), la cual participa en la degradación de las
catecolaminas DA y norepinefrina (NE). Se expresa altamente en las regiones del lóbulo frontal, donde es responsable de la regulación de los niveles de DA.
Los estudios que investigan la asociación entre COMT y TDAH se han centrado en gran medida en un SNP funcio- nal (rs4860) en el exón 4 que conduce a una sustitución de aminoácidos (valina – metionina). Se ha demostrado que este polimorfismo afecta considerablemente la actividad de la en- zima COMT, de modo que los portadores homocigotos del ale- lo valina muestran una actividad de 3 a 4 veces mayor que los portadores homocigotos del alelo metionina, lo que resulta en una menor disponibilidad de DA en los primeros (11, 25).
Corral-Frias et al., realizaron un meta análisis donde rela- cionan esta variante a alteraciones en aprendizaje por recom- pensa (26).
Otros estudios no encontraron evidencia de relación (11).
Gen DRD1
Codifica para el receptor de DAD1, siendo éste el subtipo de receptor de DA más abundante en el cerebro. Está alta- mente expresado en el cuerpo estriado y la corteza cerebral.
La variación genética común en el gen DRD1 (rs5326) se ha asociado con el riesgo de esquizofrenia y deterioro cogniti- vo en pacientes con trastorno bipolar, siendo ambas entidades comórbidas con el TDAH (27).
Gen HTR1B
Codifica para el receptor 1B de 5HT localizado en el cro- mosoma 6 (6q13), el más estudiado en TDAH. Es un receptor acoplado a proteína G que inhibe la formación de AMPc. Se expresa altamente en el núcleo del rafe dorsal, que participa en el ciclo sueño/vigilia, y en menor grado en el cuerpo estria- do y las regiones frontales, como la CPF dorsolateral. El estu- dio inicial, que investigó a 273 familias nucleares con TDAH, informó una transmisión preferencial del alelo G rs6296 a los probandos de TDAH (28).
Gen LPHN3
Codifica para el gen de latrofilina 3 (Receptor L3 acopla- do a proteína G de adhesión, ADGRL3, cromosoma 4). Am- pliamente expresado en núcleo caudado, cerebelo, amígdala y corteza cerebral. Se identificó como un gen de riesgo de TDAH después de estudios de ligamiento genético en fami- lias de casos múltiples. En estudios de asociación multisi- tio, inicialmente, se demostró que un haplotipo de tres SNP (rs6551665, rs1947274 y rs2345039) estaba asociado con el TDAH. Su deficiencia se correlaciona con alteraciones en la
expresión cortical de DAT (11, 29).
Hay modelos de ratones knockout de este gen, que reprodu- cen la signo sintomatología del TDAH (8).
Gen MAOA
Codifica para la enzima monoaminooxidasa A, que intervie- ne en la degradación de monoaminas. Se encuentra localizado en cromosoma X. Se halló que un SNP, rs1137070 (ubicado en el exón 14), contribuye a la impulsividad, puntuaciones altas de TDAH y mal resultado en los tratamientos (30).
Gen NOS1
Codifica para la enzima óxido nítrico sintasa 1, la cual sin- tetiza óxido nítrico a partir de l-arginina. El óxido nítrico es un radical libre reactivo que puede actuar como mediador biológico en varios procesos, incluida la neurotransmisión do- paminérgica y serotoninérgica y el crecimiento de neuritas. El gen tiene una estructura compleja, que incluye 12 primeros exones alternativos no traducidos (exón 1a-1l). En el exón 1f, un VNTR que afecta la expresión genética se ha relacionado con un comportamiento hiperactivo e impulsivo en humanos. Se demostró que el alelo corto es el factor de riesgo para el TDAH, especialmente en mujeres (11, 31).
Gen SERT
Codifica para el Transportador de Serotonina (sinónimos 5HTT, SLC6A4, localizado en brazo largo del cromosoma 17). Se expresa en regiones implicadas en la atención, la memoria y las actividades motoras como: amígdala, hipocampo, tála- mo, putamen y ACC. Un metaanálisis proporcionó evidencia significativa de una asociación entre el TDAH en niños y la variante larga del gen (11, 32).
En ratones DAT KO, la administración de inhibidores del transportador de serotonina o de varios agonistas y antago- nistas del receptor de 5HT (agonistas no selectivos quipazina y 5-carboxamidotriptamina o el antagonista selectivo 5HT2A M10090) reduce drásticamente la hiperactividad (33).
Gen del SNAP25
Codifica para la proteína 25 asociada al sinaptosoma. Ésta participa en el crecimiento axonal y la plasticidad sináptica, así como en el acoplamiento y fusión de vesículas sinápticas en las neuronas presinápticas (necesarias para la regulación de la liberación de neurotransmisores). Varios estudios han probado el vínculo y la asociación entre SNAP25 y el TDAH. Un metaanálisis inicial del SNP rs3746544 sugirió que SNAP25 era un gen de riesgo de TDAH (11, 3). En ratones modificados genéticamente (Coloboma mice), se demostraron alteraciones en el sistema monoaminérgico, con reducción de
la recaptación de DA en el estriado dorsal y aumento de la concentración de NE en el estriado, locus ceruleus y núcleo accumbens. La hiperactividad en estos ratones mejoró con la administración de anfetaminas, pero no con MPH (8).
Gen de la TPH2
Codifica para la enzima triptófano hidroxilasa 2, que catali- za la reacción del triptófano a 5-hidroxitriptófano, que poste- riormente se descarboxila formando el neurotransmisor 5HT. Es activa en SNC, mientras que la TPH1 lo es en sistema ner- vioso periférico. Se encontró una asociación con el TDAH para varios SNP. Para rs4570625, se demostró que el alelo G se transmite con mayor frecuencia a hijos con TDAH, lo mismo que el alelo T del rs11178997 (34).
Sheehan et al., (2005) estudiaron 8 SNP adicionales en 179 familias con TDAH y encontraron evidencia estadística- mente significativa de asociación con el alelo rs1843809-T (p=0,0006), el alelo rs1386497-A (p=0,048) y una tendencia que sugiere asociación con el rs1386493- Alelo C (p=0,09). En el proyecto IMAGE, rs1843809 y rs1386497 también se asociaron significativamente con el TDAH (35).
Gen DRD5
Codifica para el receptor dopaminérgico D5. Consta de un único exón en el cromosoma 4. Dos metaanálisis confirmaron que un microsatélite polimórfico sin importancia funcional conocida, confiere un riesgo pequeño pero significativo de TDAH. Se observó asociación entre el alelo repetido de 148 pb y 4 variables del test de variables de atención (errores de comisión, errores de omisión, tiempos de reacción y variabili- dad del tiempo de reacción) (36).
Gen ADRA2A
Codifica para el receptor adrenérgico Alpha-2A. Se informó una asociación entre el alelo G-1291C con el TDAH y los sín- tomas del trastorno de conducta o de oposición desafiante. Al mismo tiempo, el alelo C-1291G se asoció con un espectro de otras afecciones, incluidos los ataques de pánico, trastorno obsesivo compulsivo (TOC), adicciones, síntomas afectivos y esquizoides (37).
Otros genes
El gen del BDNF (cromosoma 11p13) ha sido asociado con hiperactividad, hiperfagia y obesidad en ratones (38). En hu- manos se identificó y relacionó una sustitución de valina por metionina en el codón 66 del gen (Val66Met) (25).
El gen Arsa (cromosoma 22q13.33, que codifica para la enzi- ma arilsulfatasa A) está relacionado con baja performance escolar, trastornos de conducta, ataxia cerebelosa y leucodistrofia (39).
Los modelos de ratón del gen Disc1 (Disrupted in Schi- zophrenia 1) muestran comportamientos típicos asociados a trastornos psiquiátricos como alteraciones en la actividad loco- motora (hiperactividad en machos e hipoactividad en hembras), impulsividad y aumento del comportamiento de desesperación. El gen codifica para la proteína homónima, que interviene en procesos de migración y crecimiento neuronal (40).
El gen CNR1 (receptor de cannabinoide 1) también se en- contró asociado con el trastorno en estudios de asociación basados en genes candidatos. Además, se ha observado una asociación de CNR1 con el abuso y la dependencia del alco- hol y las drogas (41).
El gen BAIAP2 (brain-specific angiogenesis inhibitor 1-as- sociated protein 2) se correlacionó con TDAH persistente. Interviene en proliferación y supervivencia neuronal, morfogé- nesis y maduración de espinas dendríticas y guía del cono de crecimiento neuronal (42).
El FOXP2 también se asoció con TDAH en adultos, como así también en trastornos del lenguaje (43).
Un estudio de 489 pacientes con TDAH y 1285 controles encontró CNV raras en el gen de la proteína 2 del Parkinson (PARK2). Ésta regula el sistema ubiquitina-proteosoma de la célula, que ayuda a eliminar las proteínas dañadas y en ex- ceso (44).
Comings en 1991, comparó a 104 sujetos con TDAH (casi todos con síndrome de Tourette comórbido) con controles y encontró una asociación significativa con el alelo TaqIA1 del gen DRD2 (Receptor de DA 2) (45).
Más adelante se mencionarán otros significativos.
Estudios de transcriptomas
El primer estudio de expresión génica mediante microarrays se publicó en 2012 y describió niveles elevados de prosta- glandina-D2-sintasa (PTGDS) en sangre periférica de 30 ni- ños con TDAH en comparación con 9 niños con TBP (46).
Otro estudio informó datos de microarrays de 45 adultos con TDAH que no habían recibido medicación y 39 contro- les sanos, mostrando sobreexpresión de RNF122 (Ring Finger Protein 122) en sujetos con TDAH. RNF122 es una ubiqui- tina ligasa E3 implicada en el procesamiento, el tráfico y la degradación de proteínas mediados por proteosomas y actúa como mediador esencial de la especificidad del sustrato de la ligadura de ubiquitina (44).
Mortimer et al., (2020) estudiaron el perfil de transcripto- ma en células mononucleares de sangre periférica a 94 perso- nas adultas con TDAH vírgenes de tratamiento farmacológico vs 124 controles sanos. Detectaron evidencia sugestiva de asociación (valor de p <0,05 antes de la corrección para prue- bas múltiples) para 1793 genes, de los cuales 48 ya estaban relacionados con TDAH en trabajos previos. A través del es- tudio exploratorio encontraron 8 genes que muestran niveles de expresión diferenciales en el TDAH, con una dirección de efecto consistente en dos conjuntos de datos independientes. Incluyen genes relacionados con la esquizofrenia y el logro educativo (KMT5A, lysine methyltransferase 5A), diferencia- ción neuronal y migración (KLF4 Kruppel like factor 4 e IL7R interleukin 7 receptor), tráfico de membranas y crecimiento de axones (RAB11FIP1, RAB11 family interacting protein 1) dependencia del alcohol (SLA, Src like adaptor), insomnio (SNORA38, small nucleolar RNA. H/ACA box 38) y la función cognitiva (EGR2, early growth response 2). Estos resultados respaldan estudios previos que revelan antecedentes genéti- cos compartidos y vías biológicas comunes que subyacen a diferentes trastornos psiquiátricos y afecciones comórbidas con TDAH (47).
En un metaanálisis sobre 17149 casos y 32411 contro- les, se encontró asociación con 9 genes: FEZF1, DUSP6, ST3GAL3/KDM4A, SEMA6D, C2orf82 / GIGYF2, AMN y
FBXL17. El primero es candidato para TEA, interviniendo en la formación del diencéfalo (48).
Weiß et al., (2021) estudiaron la metilación del ADN en 37 genes candidatos mediante la realización de una secuencia- ción dirigida con bisulfito del ADN aislado de sangre total de 88 personas diagnosticadas con TDAH adultas y 91 personas no afectadas (edad media 34,2 años). Los sitios metilados diferencialmente se evaluaron mediante modelos lineales generalizados que prueban el estado del TDAH y los sínto- mas del TDAH, teniendo en cuenta además una puntuación de tabaquismo basada en la metilación, la edad, el sexo y el recuento de células sanguíneas. La metilación del ADN de sitios únicos dentro de DRD4 y KLDR1 se asoció con el estado de TDAH en adultos, y múltiples sitios de metilación de ADN dentro de TARBP1 se asociaron con síntomas de TDAH en la edad adulta y la infancia. Este constituye el primer trabajo que apuntan a TARBP1 como un nuevo gen candidato para los síntomas del TDAH. Los niveles de metilación de TARBP1 se asocian con la gravedad de los síntomas del TDAH y el tabaquismo prenatal. En estudios ontológicos se lo relacionó a la regulación de la transcripción por la ARN polimerasa II, al procesamiento de ARN y a la metilación de ARNt. Es in- teresante que algunas variantes fueron asociadas con EZQ y psoriasis, ambas entidades comórbidas con TDAH (49).
En el año 2023 se realizó un estudio de asociación de todo el transcriptoma (TWAS) utilizando un metaanálisis del GWAS realizado por Demontis en el mismo año (50) (el cual iden- tificó 27 locus de riesgo), en 38.691 personas con TDAH y 186.843 controles, y 14 paneles de referencia de expresión genética en múltiples tejidos cerebrales y sangre total. Con base en los resultados de TWAS, seleccionaron subconjuntos de genes y establecieron puntuaciones de riesgo transcrip- tómico (TRS) para el trastorno en células mononucleares de sangre periférica de individuos con TDAH y controles. Encon- traron evidencia de asociación entre el TDAH y los TRS cons- truidos utilizando perfiles de expresión de múltiples áreas del cerebro, y los individuos con TDAH tienen una mayor carga de TRS que los controles. Los TRS no estaban correlacionados con la puntuación de riesgo poligénico (PRS) para el TDAH y, en combinación con el PRS, mejoraron significativamente la proporción de varianza explicada con respecto al modelo solo PRS. Estos resultados respaldan el potencial predictivo com- plementario de los perfiles genéticos y transcriptómicos en sangre y subrayan la utilidad potencial de la expresión genéti- ca para la predicción de riesgos y una comprensión más pro- funda de los mecanismos moleculares subyacentes al TDAH. Los genes están relacionados a transmisión glutamatérgica, neurotransmisión DA y NE y desarrollo neuronal (PNPLA2, PLK1S1, GMPPB, GIGYF2 y SLC25A22). Su detalle está fue- ra del alcance de este artículo (51).
Estudios de GWAS
Neale et al., (2008) llevaron a cabo el primer GWAS fami- liar en TDAH (estudio IMAGE) en 909 tríos de afectados, 790 varones y 119 mujeres, de 5 a 17 años. 845 presentaban TDAH subtipo combinado, siendo el promedio de síntomas de 16.1. Los participantes eran israelíes y europeos caucá- sicos. El hallazgo más consistente fue la posible implicación del gen cadherina 13 (CDH13) en el TDAH. La cadherina-13 es una proteína dependiente de calcio importante en la adhe- sión célula-célula y el crecimiento de células neurales. Este gen se expresa en la corteza cerebral y se ha relacionado con déficits de memoria de trabajo, hiperactividad e impulsividad en personas con TDAH (59). Otro gen asociado fue GFOD1 (Glucose-Fructose Oxidoreductase Domain Containing 1, en cromosoma 6), que se expresa en el cerebro y posible- mente desempeñe un papel en el transporte de electrones
(57). Otros genes candidatos hallados en ese estudio fueron: DCLK1 (organizador del citoesqueleto), SPOCK3 (componen- te de la matriz extracelular) y 2 reguladores de canales de K (KCNIP1 y KCNIP4) (52).
En un estudio se encontraron duplicaciones en genes que codifican para el receptor metabotrópico de glutamato (GRM,
mGluR): duplicaciones en GRM1 y eliminaciones en GRM-5, 7 y 8. Son fundamentales en neurogénesis (53).
Reif et al., (2009) hallaron una asociación del SNP en NOS1, que codifica la forma neuronal de la óxido nítrico sintasa. El NO actúa como segundo mensajero del receptor N-metil-D-aspartato (NMDA) e interactúa tanto con el sistema dopaminérgico como con el serotoninérgico del cerebro hu- mano. NOS1 se ha asociado con comportamiento impulsivo y agresivo y TDAH (31).
Lesch et al., (2008) publicaron un GWAS donde analizaron 343 pacientes alemanes con TDAH persistente (edad media 32,9 años con un rango de 18 a 65 años, 54,5% hombres,
61,5% subtipo combinado, 31,2% subtipo inatento, 7,3% subtipo hiperactivo-impulsivo) y 304 controles (media 32,7 años, 51,3% hombres). La comorbilidad a lo largo de la vida con depresión mayor fue del 44,7% y con trastornos por uso de sustancias 44,1% (de los cuales 32,0% por consumo de cannabis, 40,6% por consumo de alcohol). Después del análisis de datos, los autores seleccionaron frecuencias alé- licas de 504.219 SNP, de los cuales los SNP autosómicos se clasificaron según la media de las clasificaciones reali- zadas mediante ANOVA y sumas de rangos. Informan genes candidatos para el TDAH: KALRN (15 SNP), ZNF354C (15 SNP), WRNIP1 (14 SNP), GRB10 (10 SNP), DPP6 (7 SNP), ARHGAP22 (11 SNP), RAB38 (11 SNP), FAT3 (8 SNP), DA259379 (13 SNP), NT5DC3 (20 SNP), ASTN2, CSMD2,
ITGA11, CTNNA2 y CDH13. Todos estos genes han mostrado asociación con trastornos por uso de sustancias y fenotipos relacionados, lo cual es interesante a la luz de la frecuente comorbilidad con dichos trastornos en el TDAH (44,1 % en el estudio actual) (54).
Un GWAS cruzado de 5 trastornos neuropsiquiátricos prin- cipales (esquizofrenia, trastorno bipolar, TEA, trastorno de- presivo mayor y TDAH) identificó 5 hallazgos significativos en todo el genoma, cuatro de los cuales en o cerca de los genes ITIH3, AS3MT, CACNA1C y CACNB2 , fueron compartidos con TDAH (55).
En el año 2013 se publica el primer GWAS de TDAH en población china de la etnia Han, que abarcó 1.040 casos y 963 controles. Aunque no se encontró ninguna variante sig- nificativa de SNP o CNV en todo el genoma, sí se halló evi- dencia significativa de un componente poligénico de SNP y una mayor carga de CNV raras. Estas implican 3 vías: ad- hesión celular (NXPH1–NRXN1, CNTN2–CNTNAP2–ZMIZ1),
desarrollo de sinapsis glutamatérgicas (GRM7–PICK1–GRM3, PICK1–EPHA7) y vías de transcripción (TAF2–PAPOLA–POL- R2F–MED27–MED20, POLR2F – NCL–RSL1D1–BYSL) (56).
Un estudio investigó 429.784 SNP autosómicos utilizando el algoritmo FBAT-logrank para identificar variantes genéticas que podrían predisponer a los niños a una edad más temprana de aparición de los síntomas de la enfermedad. Se probaron modelos genéticos aditivos, dominantes y recesivos. Un total de 16 SNP alcanzaron valores de asociación, no existiendo superposición con los resultados de otros GWAS. Aunque po- tencialmente debido al azar, es notable la mención de una se- rie de genes que están involucrados en la respuesta del cuer- po a las exposiciones ambientales y/o regulados por el medio ambiente, como el ADAMTS2 relacionado con la inflamación, el regulador del estrés MAP3K7 y el gen NAV2 que responde a la vitamina A (57).
Klein et al., (2019) investigaron la superposición entre la variación genética común asociada con el riesgo de TDAH y medidas de volumen cerebral para identificar los procesos biológicos subyacentes que contribuyen al trastorno. Para eso tuvieron en cuenta estudios previos que encontraron dismi- nución del volumen del n accumbens, amígdala, n caudado, hipocampo y putamen.
En los niveles de variante única y de todo el gen, los metaa- nálisis encontraron locus significativos asociados tanto con el riesgo de TDAH como con los volúmenes cerebrales. Identifi- caron a los genes SEMA6D, KIZ y MEF2C como posibles loci clave que contribuyen tanto al riesgo de TDAH como a ICV, y el análisis exploratorio del conjunto de genes reveló una aso- ciación de la superposición de TDAH-ICV con la variación en los genes de crecimiento de neuritas (58).
Un GWAS-MA a gran escala en TDAH, considerando 20.183 individuos con TDAH y 35.191 controles, identificó por primera vez 12 locus significativos en todo el genoma, ubicados en o cerca de genes implicados en procesos de neurodesarrollo y fun-
ciones neurobiológicas, potencialmente relevante para el TDAH. Entre ellos, destacan FOXP2 en el locus del cromosoma 7, que está implicado en la formación de sinapsis y el desarrollo del habla y el aprendizaje así como DUSP6 (cromosoma 12), que puede jugar un papel en la regulación de la homeostasis de los neurotransmisores al afectar los niveles de DA en las sinapsis. Las otras localizaciones son: ST3GAL3, KDM4A, KDM4A-AS1, PTPRF, SLC6A9, ARTN, DPH2, ATP6V0B, B4GALT2,
CCDC24, IPO13 (en cromosoma 1), SPAG16 (cromosoma 2), PCDH7, LINC02497 (cromosoma 4), LINC00461, MIR9–2, LINC02060, TMEM161B-AS1 (cromosoma 5), LINC01288
(cromosoma 8), SORCS3 (cromosoma 10), SEMA6D (cromoso- ma 15) y LINC01572 (cromosoma 16) (13, 59).
En el año 2021, Liu et al., presentaron el primer estudio que utilizó una red neuronal convolucional para clasificar 1033 per- sonas diagnosticadas con TDAH (870 varones, 84,2%) y 950 controles sanos (601 varones, 63,3%) según sus datos genó- micos (Ver Cuadro 3). Al incorporar el análisis de prominencia de la red de aprendizaje profundo (Deep learning model), se encontró un total de 96 genes candidatos, de los cuales 14 se han informado en estudios anteriores relacionados con el TDAH. Además, el análisis conjunto de locus de rasgos cuan- titativos de expresión y enriquecimiento de ontología genética identificó un gen de riesgo potencial para el TDAH, EPHA5 (ephrin type-A receptor 5, cromosoma 4q13) con una variante de rs4860671. Ephrin A5 es una proteína anclada a glicosil- fosfatidilinositol (GPI) de la subclase efrina-A de ligandos de efrina que participa en el desarrollo del cerebro, la remodela- ción sináptica y la plasticidad sináptica en el cerebro adulto a través de la regulación de la sinaptogénesis. Otros genes in- volucrados incluyeron NRG3, TENM4, LIG4, MDGA2, BMP2, EPHA5, EPHA7, LPAR1 y TLR (60).
Un metaanálisis de cuatro estudios de GWAS en el TDAH
Cuadro 3
Red neuronal convolucional y deep learning (Definiciones)
| Red neuronal convolucional (CNN o ConvNet) es una arquitectura de red para Deep Learning que aprende directamente a partir de datos. Son particularmente útiles para identificar patrones en imágenes con el fin de reconocer objetos, clases y categorías. |
| Deep Learning es un subconjunto del machine learning, que es esencialmente una red neuronal con tres o más capas. Estas redes neuronales intentan simular el compor- tamiento del cerebro humano, lo que les permite “aprender” de grandes cantidades de datos. |
sugirió que el CHMP7 (charged multivesicular body protein, localizado en el cromosoma 8p21.3 y participa en el trans- porte tardío del endosoma a la vacuola y la división nuclear mitótica) es un gen predisponente. Se ha demostrado que una variante de ADN (rs2294123) asignada a CHMP7 tiene una alta probabilidad de funcionalidad y se correlaciona con nive- les reducidos de transcripción. Se correlacionaría con buena respuesta al MPH (61).
Recientemente, Hongyao et al., (2023) realizaron un es- tudio de asociación de red de genes entre TDAH y dislexia, entidades que presentan una alta comorbilidad (25 al 48%). Hallaron que entre los 20 genes con mayor prioridad DRD2, DRD4, CNTNAP2 (relacionado a encefalopatía epiléptica) y GRIN2B (que codifica para una proteína que forma parte de los receptores ionotrópicos de tipo NMDA), se mencionan en la literatura como directamente relacionados con la comorbi- lidad del TDAH y la dislexia. Además los genes OPRM1 (gen del receptor opioide µ1), CHRNA4 (codifica para el receptor nicotínico alfa 4 ) y el SNCA (proteína alfa-sinucleína) se des- cribieron como candidatos noveles en el desarrollo de comor- bilidad de estas dos enfermedades. Los genes mencionados se relacionan, con la transducción de señales, regulación de la transcripción de los promotores de la ARN polimerasa II, transmisión sináptica química, respuesta a fármacos, trans- porte transmembrana de iones, desarrollo del sistema nervio- so, adhesión celular y migración neuronal (62).
En el año 2022, Ajnakina et al., utilizando datos del Es- tudio Longitudinal Inglés sobre el Envejecimiento (ELSA), (estudio multidisciplinario en curso de una muestra nacio- nalmente representativa de la población inglesa mayor de 50 años), encontraron un riesgo elevado de mortalidad relacio- nado a la predisposición multigénica del TDAH, utilizando el Score Poligénico (PGS), relacionado a asma, EPOC, DBT 2 y elevado riesgo cardiovascular, independientemente de las conductas de riesgo conocidas en esta población (consumo de sustancias, tabaquismo, conductas delictivas, accidentes). El riesgo de EPOC se correlacionó con menor edad de la madre al momento del parto (teniendo en cuenta que los embarazos adolescentes son más frecuentes en el grupo de riesgo (63).
El New Jersey Language and Autism Genetics Study (NJLAGS) recopiló más de 100 familias con al menos un miembro afectado por TEA. Estas mostraron una alta preva- lencia de TDAH, brindando una buena oportunidad para estu- diar los factores de riesgo genéticos compartidos para el TEA y el TDAH. El estudio de vinculación de las familias NJLAGS reveló regiones en los cromosomas 12 y 17 que están sig- nificativamente asociadas con el TDAH. Utilizando datos de secuenciación del genoma completo en 272 muestras de 73
familias NJLAGS, identificaron genes de riesgo potenciales para ambas entidades. Dentro de las regiones de ligamiento, hay 36 genes asociados con el TDAH mediante un enfoque de priorización de genes basado en pedigrí. KDM6B (lisina des- metilasa 6B) es el gen de mayor rango, siendo un gen de ries- go conocido para trastornos del desarrollo neurológico. Otros fueron MYBBP1A, PER1 y TIMELESS, todos relacionados al ritmo circadiano (64).
Discusión
La etiología del TDAH es compleja y multifactorial. La teo- ría más plausible hasta la fecha es que surge de múltiples factores de riesgo genéticos y ambientales, que tienen peque- ños efectos individuales y actúan en conjunto para aumentar la susceptibilidad a desarrollar el trastorno. Esto constituye un patrón de herencia no mendeliana, compleja, con posible penetrancia incompleta y expresividad variable, que sugiere la acción conjunta de múltiples genes de efecto moderado o discreto con factores ambientales.
Otra hipótesis que se debe explorar en futuras investigacio- nes es la posibilidad de que el TDAH sea un trastorno omni- génico. El modelo omnigénico de Boyle et al., (65) postula la existencia de una pequeña cantidad de genes centrales que tienen papeles biológicamente interpretables en la enferme- dad junto con una cantidad mucho mayor de genes periféricos que regulan a estos genes centrales.
La evidencia actual iría a favor de que los casos de TDAH pueden definirse como extremos de la distribución de los sín- tomas del mismo (inatención /hiperactividad/impulsividad) y que los subtipos no corresponderían a distintas genéticas. Los estudios de GWAS sobre estos rasgos revelaron influencias genéticas tanto únicas como compartidas en las dimensiones del TDAH, en sintonía con la tendencia actual del concepto de espectro. Dentro de esto debemos considerar la fuerte evi- dencia de las relaciones genéticas y fenotípicas entre TDAH y TEA, y la comorbilidad con múltiples trastornos, como el consumo de sustancias, trastornos del humor y ansiedad, tras- torno antisocial, asma, DBT, EPOC y obesidad entre otros.
Quedan fuera de este trabajo los importantes aportes de la farmacogenómica, estudios detallados sobre correlación estructura-función (medidas mediante neuroimágenes fun- cionales y/o test neuropsicológicos)-genética y las intrincadas interacciones GxA.
Todo esto refleja la complejidad del tema y la puesta en evidencia de que cuanto más sabemos de genética, más im- portancia le debemos dar al ambiente.
Referencias bibliográficas
- 1. Angemi J (2017). Diagnóstico y trata- miento del trastorno por déficit de atención e hiperactividad. En PROAPSI (Programa de actualización en psiquiatría) Barembaum R et al. Bs As. Ed. Médica Panamericana.
- 2. Nikolas M, Burt S.A. (2010). Genetic and environmental influ- ences on ADHD symptom dimensions of inattention and hyper- activity: a meta-analysis. J. Abnorm. Psychol. 119 (1), 1.
- 3. Faraone S, Larsson H (2019). Genetics of attention deficit hyperactivity disorder. Mol. Psychiatry 24 (4), 562–575.
- 4. Levy F, Hay D, Mc Stephen M, Wood C, Waldman I (1997). Attention-deficit hype- ractivity disorder: a category or a continuum? Genetic analysis of a large-scale twin study.
J. Am. Acad. Child Adolesc. Psychiatry 36, 737–744.
- 5. Sprich S, Biederman J, Crawford H, Mundy E, Faraone S. (2000). Adoptive and biological families of children and adoles- cents with ADHD. J Am Acad Child Adolesc Psychiatry; 39:1432–7.
- 6. McLoughlin G, Ronald A, Kuntsi J, As- herson P, Plomin R. (2007). Genetic support for the dual nature of attention deficit hype- ractivity disorder: substantial genetic overlap between the inattentive and hyperactive-im- pulsive components. J Abnorm Child Psychol; 35:999–1008.
- 7. Pettersson E, Anckarsater H, Gillberg C, Lichtenstein P. Different neurodevelop- mental symptoms have a common genetic etiology. (2013) J Child Psychol Psychiatry; 54:1356–65.
- 8. Cabana-Dominguez J, Anton-Galindo E, Fernandez- Castillo N, Singgih E, O’Leary A, Norton W et al (2023) The translational genetics of ADHD and related phenotypes in model organisms. Neuroscience and Biobe- havioral Reviews 144 (2023) 104949.
- 9. Matrisciano F, Tueting P, Dalal I, Kadriu B, Grayson D, Davis J et al. (2013). Epigene- tic modifications of GABAergic interneurons are associated with the schizophrenia-li- ke phenotype induced by prenatal stress in mice. Neuropharmacology 68, 184–194.
- 10. Hawi Z, Cummin, T, Tong J, Johnson B, Lau R, Samarrai W, Bellgrove M (2015). The molecular genetic architecture of attention deficit hyperactivity disorder. Mol. Psychiatry 20 (3), 289–297.
- 11. Klein M, Onnink M, van Donkelaar M, Thomas Wolfers T, Harich B, Shi Y et al (2017). Brain imaging genetics in ADHD and beyond – Mapping pathways from gene
to disorder at different levels of complexity. Neuroscience and Biobehavioral Reviews 80; 115–155.
- 12. Zhou K, Dempfle A, Arcos-Burgos M, Bakker S, Banaschewski T, Biederman J et al. (2008). Meta-analysis of genome-wide linkage scans of attention deficit hyperactivi- ty disorder. Am J Med Genet B Neuropsychia- tr Genet; 147B:1392–8.
- 13. Demontis D, Walters R, Martin J, Ma- ttheisen M, Als T, Agerbo E et al (2019). Discovery of the first genome-wide significant risk loci for attention deficit/hyperactivity di- sorder. Nat. Genet. 51 (1), 63–75.
- 14. Brookes K, Xu X, Chen W, Zhou K, Neale B, Lowe N et al (2006). The analysis of 51 genes in DSM-IV combined type atten- tion deficit hyperactivity disorder: association signals in DRD4, DAT1 and 16 other genes. Mol. Psychiatry 11,934–953.
- 15. Franke B, Vasquez A, Johansson S, Hoogman M, Romanos J, Boreatti-Hummer A et al. (2010). Multicenter analysis of the SLC6A3/DAT1 VNTR haplotype inpersistent ADHD suggests differential involvement of the gene in childhood and persistent ADHD. Neuropsychopharmacology 35, 656–664.
- 16. Da Silva M, Cordeiro Q, Louza M, Valla- da H (2009). Association between a SLC6A3 intron 8 VNTR functional polymorphism and ADHD in a Brazilian sample of adult patients. Rev Bras Psiquiatr. 2009; 31(4):387-95.
- 17. Szobot C, Roman T, Hutz H, Genro J, Shih M, Hoexter M et al (2011). Molecular imaging genetics of methylphenidate respon- se in ADHD and substance use comorbidity. Synapse 65, 154–159.
- 18. de la Peña J, de la Peña I, Custodio R, Botanas C, Kim H et al (2018). Exploring the validity of proposed transgenic animal models of attentiondeficit hyperactivity di- sorder (ADHD). Mol. Neurobiol. https://doi. org/10.1007/s12035-017-0608.
- 19. Jaber M, Dumartin B, Sagné C, Haycock J, Roubert C, Giros B et al (1999). Differen- tial regulation of tyrosine hydroxylase in the basal ganglia of mice lacking the dopamine transporter. Eur J Neurosci 11:3499–3511.
- 20. Leo D, Gainetdinov R (2013). Transge- nic mouse models for ADHD. Cell Tissue Res (354:259–271).
- 21. Rothhammer P, Paz Lagos L, Espi- nosa-Parrilla Y, Aboitiz F, Rothhammer F (2012). Variación de alelos del gen receptor de dopamina DRD4 en escolares chilenos de diferente origen étnico y su relación con ries- go de déficit atencional/hiperactividad. Rev Med Chile; 140: 1276-1281.
- 22. Nikolaidis A, Gray J (2010). ADHD and
the DRD4 exon III 7-repeatpolymorphism: an international meta-analysis. Soc. Cogn. Affect. Neurosci. 5,188–193.
- 23. Schweren L, de Zeeuw P. Durston S (2010). MR imaging of the effects ofmethyl- phenidate on brain structure and function in- attention-deficit/hyperactivity disorder. Eur. Neuropsychopharmacol. 23,1151–1164.
- 24. Rubinstein M, Phillips J, Bunzow R, Falzone L, Dziewczapolski G, Zhang G et al. (1997). Mice lacking dopamine D4 receptors are supersensitive to ethanol, cocaine, and methamphetamine. Cell 90:991–1001.
- 25. Chen J, Lipska B, Halim N, Matsumo- to M, Melhem S, KolachanaB et al. (2004). Functional analysis of genetic variation inca- techol-O-methyltransferase (COMT): effects on mRNA, protein, and enzymeactivity in postmortem human brain. Am. J. Hum. Ge- net. 75, 807–821.
- 26. Corral-Frias N, Pizzagalli D, Carre J, Mi- chalski L., Nikolova Y, Perli R et al (2016). COMT val met genotype is associated with reward learning: a replication study and meta-analysis. Genes Brain Behav. 15 (5),
503–513.
- 27. Zhao L, Lin Y, Lao G, Wang Y, Guan L., Wei J (2015). Association study of dopami- ne receptorgenes polymorphism with cogni- tive functions in bipolar I disorder patients. J.Affect. Disord. 170, 85–90.
- 28. Hawi Z, Cummins T, Tong J, Johnson B, Lau R, Samarrai W et al (2002). The mo- lecular genetic architecture of attention defi- cit hyperactivity disorder. Mol. Psychiatry 20 (3), 289–297.
- 29. Arcos-Burgos M, Jain M, Acosta M, Shi- vely S, Stanescu H, Wallis D et al. (2010). A common variant of the latrophilin 3 gene, LPHN3, confers susceptibility to ADHD and predicts effectiveness of stimulant medica- tion. Mol. Psychiatry 15, 1053–1066.
- 30. Liu L, Guan L, Chen Y, Ji N, Li H, Li Z et al. (2011). Association analyses of MAOA in Chinese Han subjects with attention-defi- cit/hyperactivity disorder: family-based asso- ciation test, case-control study, and quantita- tive traits of impulsivity. Am.J. Med. Genet. B Neuropsychiatr. Genet. 156B, 737–748.
- 31. Reif A, Jacob C, Rujescu D, Herterich S, Lang S, Gutknecht L et al. (2009). In- fluence offunctional variant of neuronal nitric oxide synthase on impulsive behaviors inhu- mans. Arch. Gen. Psychiatry 66, 41–50.
- 32. Gizer I, Ficks C, Waldma, I (2009). Can- didate gene studies of ADHD: ameta-analytic review. Hum. Genet. 126, 51–90.
- 33. Gainetdinov R, Wetsel C, Jones S, Levin D, Jaber M, Caron M (1999). Role of serotonin in
the paradoxical calming effect of psychostimu- lants on hyperactivity. Science 283:397–401.
- 34.- Walitza S, Renner T, Dempfle A, Kon- rad K, Wewetzer C, Halbach A et al. (2005). Transmission disequilibrium of polymorphic variants in the tryptophan hydroxylase-2 gene in attention-deficit/hyperactivity disorder. Mol. Psychiatry 10, 1126–1132.
- 35. Sheehan K, Lowe N, Kirley A, Mu- llins C, Fitzgerald M, Gill M, Hawi Z (2005). Tryptophan hydroxylase 2 (TPH2) gene va- riants associated with ADHD. Mol Psychia- try;10(10):944-9.
- 36. Kebir O,Tabbane K, Sengupta S, Joober R (2008). Candidate genes and neuropsycho- logical phenotypes in children with ADHD: review of association studies. J Psychiatry Neurosci; 34(2):88-101.
- 37. Comings D, Gonzalez N, Cheng Li S (2003). A “line item” approach to the iden- tification of genes involved in polygenic be- havioral disorders: the adrenergic alpha2A (ADRA2A) gene. Am J Med Genet B Neu- ropsychiatr Genet; 118B:110-4.
- 38. Kernie S, Liebl D, Parada L (2000). BDNF regulates eating behavior and locomotor activity in mice. EMBO J. 19, 1290–1300.
- 39. Lugowska A, Mierzewska H, Bekiesins- ka-Figatowska M, Szczepanik E, Goszczans- ka-Ciuchta A, Bednarska-Makaruk M (2014). A homozygote for the c.459+1G>A mutation in the ARSA gene presents with cerebellar ataxia as the only first clinical sign of metachromatic leukodystrophy. J. Neurol. Sci. 338, 214–217.
- 40. Gómez-Sintes R, Kvajo M, Gogos J, Lu- cas J (2014). Mice with a naturally occurring DISC1 mutation display a broad spectrum of behaviors associated to psychiatric disorders. Front. Behav. Neurosci. 8, 253.
- 41. Lu T, Ogdie N, Jarvelin R, Moilanen I, Loo S, McCracken J et al.(2008). Associa- tion of the cannabinoid receptor gene (CNR1) with ADHD and post-traumatic stress disor- der. Am J Med Genet B Neuropsychiatr Genet 147B:1488–1494.
- 42. Bonvicini C, Faraone SV, Scassellati C(2016). Attention-deficit hyperactivity disor- der in adults: a systematic review and metaa- nalysis of genetic, pharmacogenetic and bio- chemical studies. Mol Psychiatry; 21:1643.
- 43. Enard W, Gehre S, Hammerschmidt K, Holter S, Blass T,Somel M et al.(2009). A hu- manized version of Foxp2 affects corticobasal ganglia circuits in mice. Cell;137:961–71.
- 44.García-Martínez I, Sánchez-Mora C, So- ler Artigas M, Rovira, P, Pagerols M, Corra- les M (2017). Gene-wide association study reveals RNF122 ubiquitin ligase as a novel susceptibility gene for attention deficit hype-
ractivity disorder. Sci. Rep. 7 (1), 1–13.
- 45. Comings D, Comings G,Muhleman D, Dietz G, Shahbahrami B, Tast D(1991). The dopamine D2 receptor locus as a mo- difying gene in neuropsychiatric disorders. Journal of the American Medical Associa- tion;266(13):1793–1800.
- 46. Marín-Méndez J, Patiño-García, A. Segu- ra V, Ortuño F, Gálvez M, Soutullo C (2012). Differential expression of prostaglandin D2 synthase (PTGDS) in patients with attention deficit-hyperactivity disorder and bipolar di- sorder. J. Affect. Disord. 138 (3), 479–484.
- 47. Mortimer N, Sanchez-Mora C, Rovira P, Vi- lar-Ribó L, Richarte V, Corrales M et al. (2020). Transcriptome profiling in adult attention-defi- cit hyperactivity disorder. European Neuropsy- chopharmacology (2020) 41, 160–166.
- 48- Rovira P, Demontis D, Sánchez-Mora C, Zayats T, Klein M, Mota N et al. (2020). Shared genetic background between children and adults with attention deficit/hyperac- tivity disorder. Neuropsychopharmacology, 45:1617–1626
- 49. Weiß A, Meijer M, Budeus B, Pauper M,Hakobjan M, Groothuismink J, et al. (2021). DNA methylation associated with persistent ADHD suggests TARBP1 as novel candidate. Neuropharmacology 184 (2021) 108370.
- 50. Demontis D, Walters G, Athanasiadis G, Walters R, Therrien K, Nielsen T et al. (2023). Genome-wide analyses of ADHD identify 27 risk loci, refine the genetic ar- chitecture and implicate several cognitive domains. Nat Genet, 55:198–208.
- 51. Cabana-Domínguez J, Llonga N, Arribas L, Alemany S, Vilar-Ribó L, Demontis D et al. (2023). Transcriptomic risk scores for atten- tion deficit/hyperactivity disorder. Mol Psych
,28:3493–3502.
- 52. Neale B, Lasky-Su J, Anney R, Franke B, Zhou K, Maller Jet al. (2008). Geno- me-wide association scan of attention deficit hyperactivity disorder. Am Journal of Medi- cal Genetics Part B-Neuropsychiatric Gene- tics147B(8):1337-1344.
- 53. Elia J, Glessner J, Wang K, Takahashi N, Shtir C., Hadley et al.(2012). Genome-wi- de copy number variation study associates metabotropic glutamate receptor gene ne- tworks with attention déficit hyperactivity di- sorder. Nat. Genet. 44, 78–84.
- 54. Lesch K, Timmesfeld N, Renner J, Hal- perin R, Roser C, Nguyen T et al. (2008). Moleculargenetics of adult ADHD: converging evidence from genome-wide association and extended pedigree linkage studies. J Neural Transm 115:1573– 1585.
- 55. Cross-Disorder Group of The Psychiatric
Genomics Consortium, 2013.Identification of risk loci with shared effects on five major psychiatricdisorders: a genome-wide analy- sis. Lancet 381, 1371–1379.
- 56. Yang L, Neale B, Liu L, Lee S, Wray N, Ji N, et al. (2013). Psychiatric GWAS Con- sortium: ADHD Subgroup. Polygenic Trans- mission and Complex Neuro Developmental Network for Attention Deficit Hyperactivity Disorder: Genome-Wide Association Study of Both Common and Rare Variants. Am J Med Genet Part B 162B:419–430.
- 57. Lasky-Su J, Anney J, Neale M, Franke B, Zhou K, Maller B et al. (2008). Geno- me-wide association scan of the time to on- set of attention deficit hyperactivity disorder. Am J Med Genet B Neuropsychiatr Genet 147B:1355–1358.
- 58. Klein M, Walters R, Demontis D, Stein J, Hibar D et al. (2019). Genetic Markers of ADHD-Related Variations in Intracranial Volu- me Am J Psychiatry; 176(3): 228–238.
- 59. Pujol-Gualdo N, Sánchez-Mora C, Ramos-Quiroga J, Ribasés M, Soler Artigas M(2021). Integrating genomics and trans- criptomics: Towards deciphering ADHD. Eu- ropean Neuropsychopharmacology 44 ,1–13.
- 60. Liu L, Feng X, Li H, Li S, Qian Q, Wang Y (2021). Deep learning model reveals po- tential risk genes for ADHD, especially Ephrin receptor gene EPHA5. Briefings in Bioinfor- matics, 22(6), 2021, 1–11.
- 61. Dark C, Williams C, Bellgrove M, Hawi Z,Bryson-Richardson R(2020). Functional validation of CHMP7 as an ADHD risk gene. Translational Psychiatry 10:385.https://doi. org/10.1038/s41398-020-01077-w
- 62. Hongyao H, Chun J, Xiaoyan G, Fan- gfang L, Jing Z, Lin Z et al. (2023). Asso- ciative gene networks reveal novel candidates important for ADHD and dislexia comorbidity. BMC Medical Genomics 16:208 https://doi. org/10.1186/s12920-023-01502-1.
- 63. Ajnakina O, Shamsutdinova D, Wim- berley T, Dalsgaard S, Steptoe A(2022). High polygenic predisposition for ADHD and a greater risk of all-cause mortality: a large population-based longitudinal study. Medi- cine (2022) 20:62 https://doi.org/10.1186/ s12916-022-02279-3.
- 64.Zhou A, Cao X, Vaidhyanathan Mahaga- napathy V, Azaro M, Gwin C, Wilson S et al. (2023). Common genetic risk factors in ASD and ADHD co-occurring families. Hum Genet; 142(2): 217–230.
- 65. Boyle E, Li Y, Pritchard J (2017). An expanded view of complex traits: from polyge- nic to omnigenic. Cell. 2017; 169:1177–86.







No comments! Be the first commenter?